wiley 不允许爬虫,请勿使用下面的内容(2018.12.24)。
春去冬来,又是一年年底,公司每年都会整理相关仪器的文献,重头戏是 LI-6400,因为文献量大,需求高(LI-6800近两年开售,虽然研究成果渐渐开始发表,但是整理起来就毫不费力,总共也就 20 几篇)。于是乎出差夜里无聊的我决定用爬虫整理一下,断断续续利用出差晚上的时间琢磨了一下,历时一个多月,终于完成了。不要问我为什么耗时这么久,我想静静……,下面简单记录一下爬虫的过程吧。
工具的选择
爬虫工具选择了
Python,因为 R 下我用着趁手的确实没有,好吧,我承认我对rvest不熟悉,貌似功能也不如BeatifulSoup4多,虽然它是基于BeatifulSoup4写的。浏览器选择了
chrome,一开始用了 SelectorGadget,但后来发现不如直接用 Chrome 自带的检查网页源码的工具方便。
爬取对象
我选取了 www.onlinelibrary.wiley.com,不要问我为什么不爬谷歌的,其实我很想,但对我来讲可能只剩肉身翻墙一条路,但我目前翻不过去。至于为什么不爬百度,这个我倒是可以回答一下,不用和谷歌比,和必应的搜索结果比较一下,你就能发现答案。于是我盯上了 Wiely,主要就是他的数据库非常棒,文献质量也很好,都是英文的,不会出来个西班牙语葡萄牙语之类的,我相信在座的各位对这类文献也不感兴趣。
爬取的过程
网址
我搜索的关键词是 LICOR 及 LI-6400,直奔所需,当然,可能会有少量文献不对,但我没有核对过,搜索后共有 469 篇文献。搜索后按时间排列,发现搜索网址变为了:
随便点下方的页码后,发现每页的页码不同,例如第二页和第三页的分别为:
从中不难发现多了的部分为:&startPage= 后变化的页码以及不变的默认的每页文献数量&pageSize=20,我们利用这个来构建所有的爬取页面。
爬取的对象
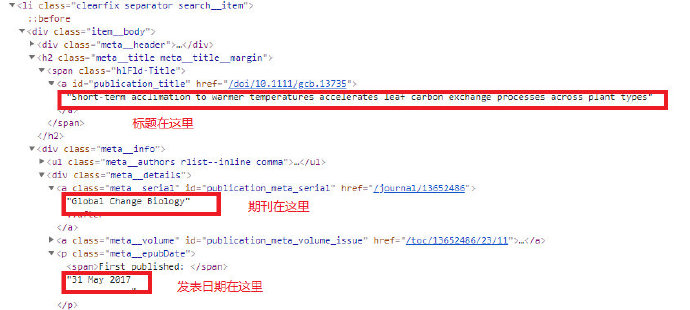
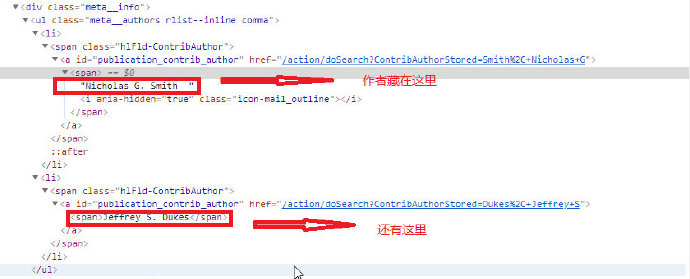
目前爬取的对象有作者、标题、期刊、发表日期及 DOI,使用发表日期不使用卷刊号是因为最新的一些文献还没见刊,附上 DOI 是因为如果有下载全文的需求而没有缴费,在不介意使用 scihub 的情况下比较方便。
所有的要爬取的内容均在一个 选择器为 class_=item__body 对 div 标签下,可以通过构建一个每个页面下的 for 循环来提取内容(下图),各部分内容对应的选择其可查看下文代码获得。
具体实现过程见代码及注释:
import requests
from bs4 import BeautifulSoup
from bs4 import SoupStrainer
import pandas as pd
import html
# element need extract stored as list
all_author = []
all_title = []
all_journal = []
all_volume = []
all_date = []
all_doi = []
all_abstract = []
# construct urls of all the pages
url = 'https://www.onlinelibrary.wiley.com/action/doSearch?AllField=li-6400%20licor%20photosynthesis&sortBy=Earliest'
headers = {'User-Agent': 'Mozilla/5.0 (Windows NT 10.0; WOW64) AppleWebKit/537.36 (KHTML, like Gecko) Chrome/69.0.3497.92 Safari/537.36'}
num = [i for i in range(25)]
page_list = [url + "&pageSize=20&" + "startPage=" + str(i) for i in num]
for each_url in page_list:
# just make sure all 24 pages are parsed
print(each_url)
r = requests.get(each_url,headers=headers)
soup = BeautifulSoup(r.text, "lxml")
# all the data are in the tag div item__body, loop it in the page
for each in soup.find_all('div', class_="item__body"):
# all the title in this tag, use get_text to extract all the text of <a>
title = each.find('a', id='publication_title').get_text()
title = html.unescape(title)
all_title.append(title)
# doi all so in this selector, as href
doi = each.find('a', id='publication_title').get('href')
# doi have https, it is wrong to extact from href
all_doi.append(doi.replace("/doi/", "https://doi.org/"))
# all the author in ul of selector meta__...
author_space = each.find('ul', class_='meta__authors rlist--inline comma').get_text()
author = author_space.strip().replace('\n', ', ').replace(" ,", ",")
author = html.unescape(author)
all_author.append(author)
journal = each.find('a', id='publication_meta_serial').get_text()
journal = html.unescape(journal)
all_journal.append(journal)
date = each.find('p', class_='meta__epubDate').get_text().strip().replace("First published: ", "")
all_date.append(date)
if each.find('a', id='publication_meta_volume_issue'):
volume = each.find('a', id='publication_meta_volume_issue').get_text()
else:
volume = "online"
all_volume.append(volume)
if each.find ('div', class_='article-section__content abstractlang_en main'):
abstract = each.find('div', class_='article-section__content abstractlang_en main').get_text()
abstract = html.unescape(abstract).strip()
elif each.find('div', class_='article-section__content main'):
abstract = each.find('div', class_='article-section__content main').get_text()
abstract = html.unescape(abstract).strip()
else:
abstract = "a"
all_abstract.append(abstract)
table = {"Author": all_author, "Title": all_title, "Journal": all_journal,
"Volume": all_volume, "Date": all_date, "Doi": all_doi}
ab_tab = {"Abstract": all_abstract}
wiley_data = pd.DataFrame(table)
wiley_abs = pd.DataFrame(ab_tab)
wiley_data.to_csv("wiley_data.csv")
wiley_abs.to_csv("wiley_abs.csv")标题词云
我们简单看一下所有文章标题的词云:

结果除了我们不意外的一些词汇外(photosynthesis,leaf 等),我们看到,做水稻的人发表的光合文章是非常多的,胁迫研究,模型等也不少。
最终结果
最终结果如下,可能有少部分人名乱码(例如德语,西班牙语等),目前网页显示还无法解决,因为我导出时已经使用了 utf-8 编码,转成的 txt 文件是没有问题的。
后记:乱码问题解决,是excel 默认的编码是 ASNI,而我们使用的 utf-8,因此乱码,解决方法是不要直接用 excel 打开,二是用数据的 “自文本” 导入,同时在弹出的界面选择 “分隔符号”后,在下面的 “文件原始格式”,选择 “65001: unicode(utf-8)”,这样打开之后就不再有问题。
转为 bib 格式
转为 bib 格式比较简单,因为 R 中有一个神器的软件包叫做 rcrossref,利用 doi 生成各种格式:
library(rcrossref)
library(stringr)
wiley <- read.csv("wiley_data.csv", stringsAsFactors = FALSE)
dois <- wiley$Doi
dois <- str_replace_all(dois, "https://doi.org/", "")
ref <- lapply(dois, cr_cn, format = "bibtex", style = "apa")
readr::write_lines(ref, "wileyonline.txt")